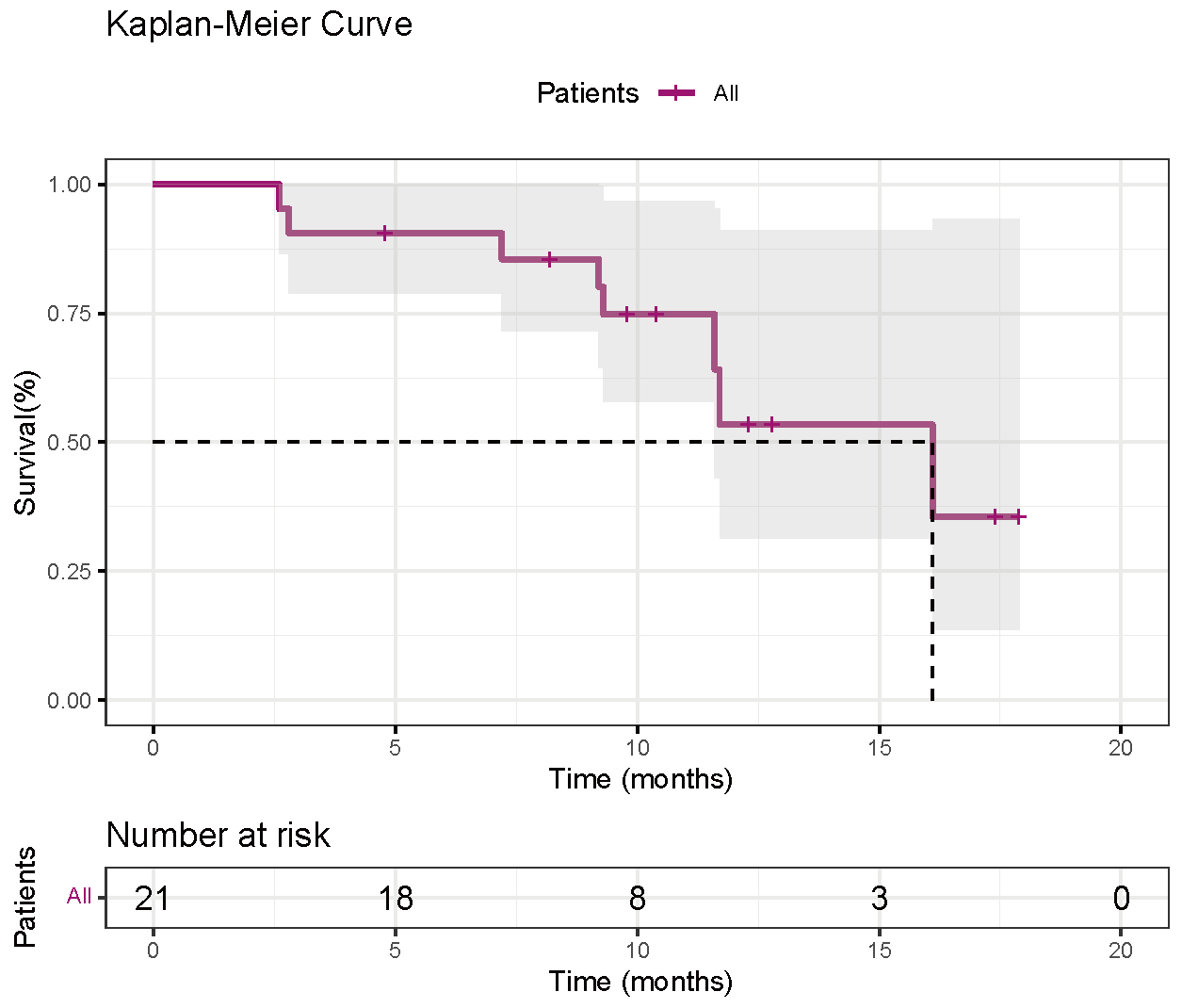
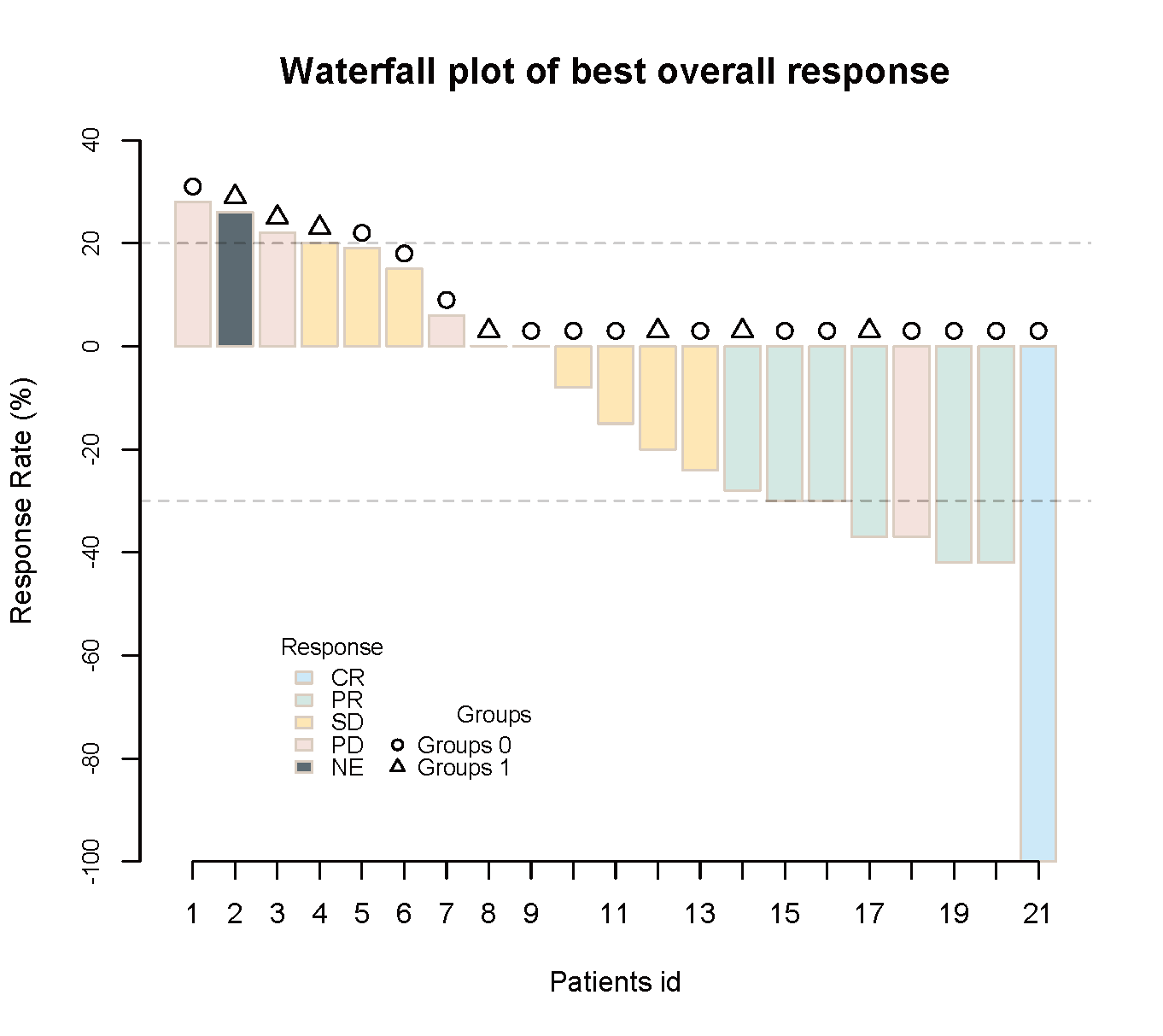
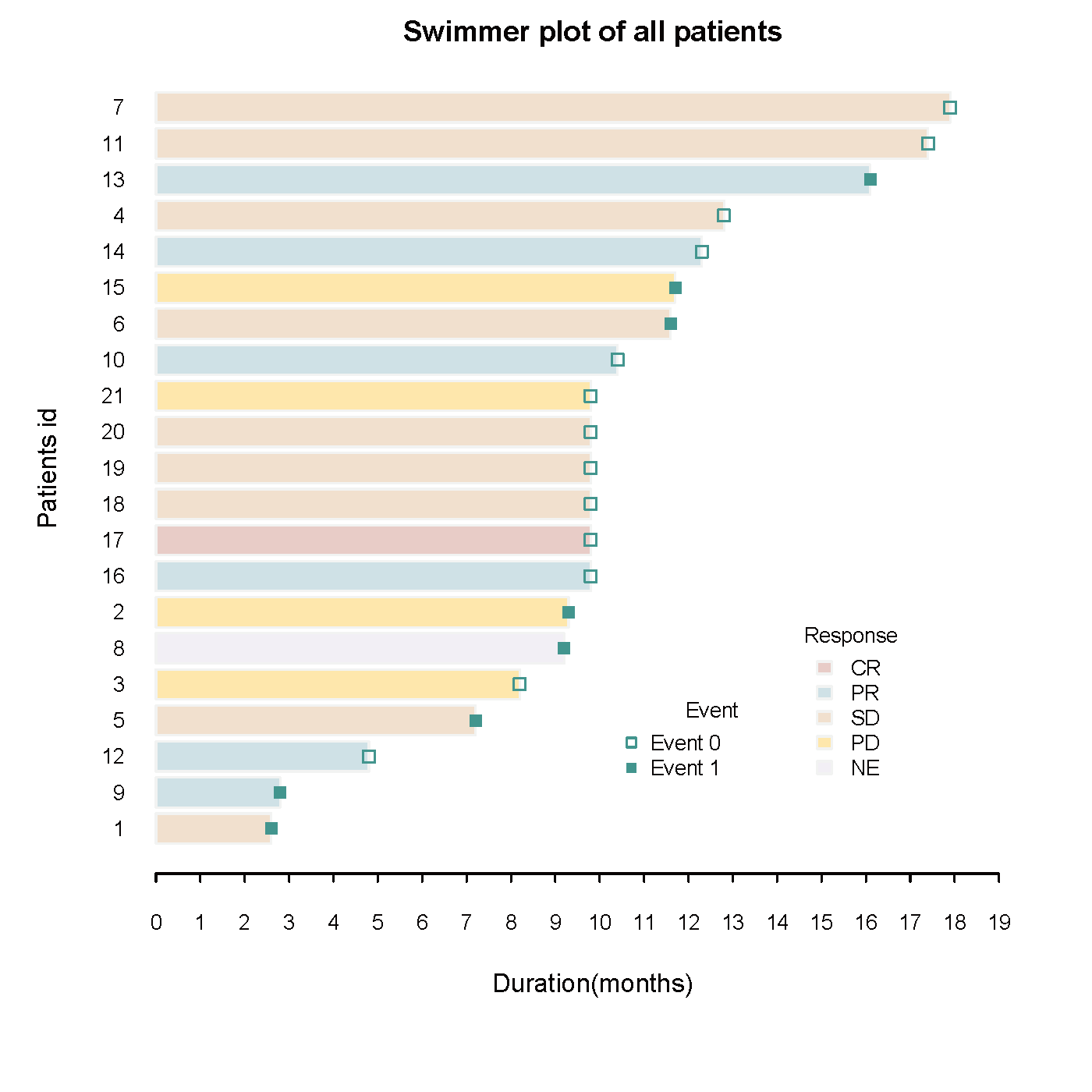
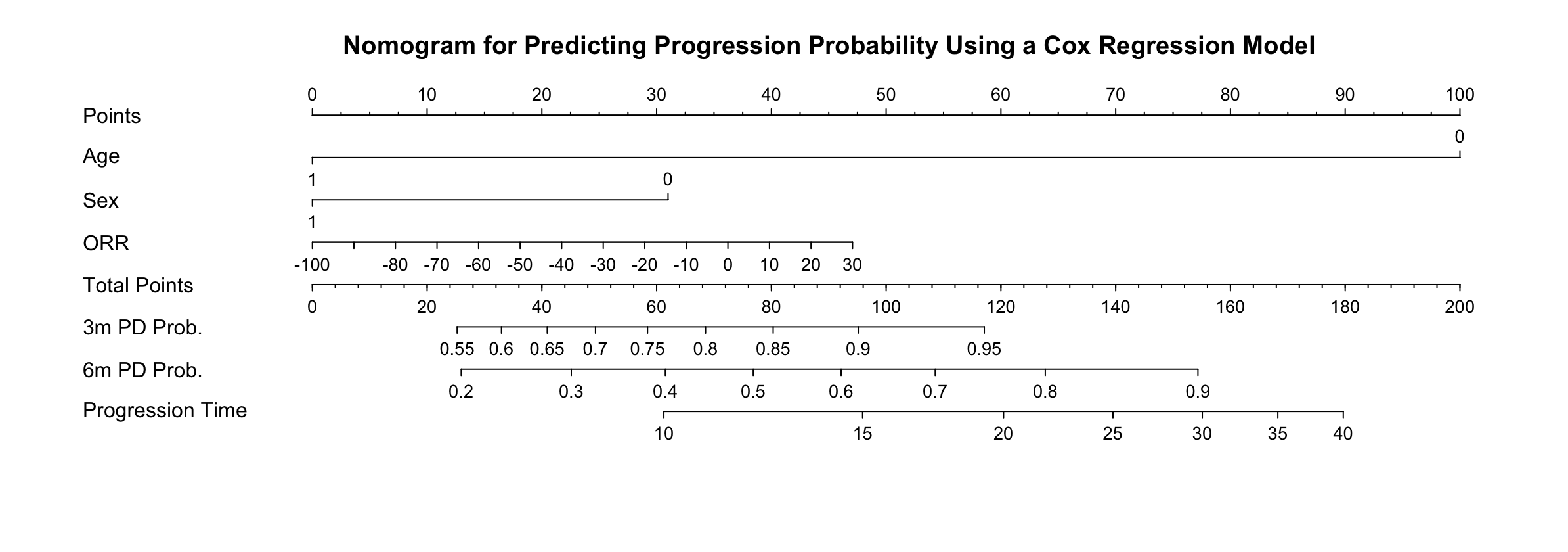
连续变量的分组均数/中位数比较(组别≥2)
基于以下规则,进行均数/中位数比较流程:
-
独立性假设
-
正态分布假设 Shapiro-Wilk test (n≤50) 或 Kolmogorov-Smirnov test (n>50)
如果不符合正态分布,采用非参数检验,对中位数采用 Mann-Whitney U 检验(2组)或 Kruskal-Wallis 检验(多组);
如果符合正态分布,进入下一步。
-
方差齐性假设
如果方差齐,且为两组,对均数进行t检验;如果为三组或更多,对均数进行F检验(单因素方差分析)。
如果方差不齐,且为两组,对均数进行Welch's t检验;如果为三组或更多,对中位数采用Kruskal-Wallis检验。